Os pesquisadores de todo o mundo trabalham para entender como o vírus da síndrome respiratória aguda grave - coronavírus 2 (SARS-CoV-2) interage com o hospedeiro humano.
O genoma do SARS-CoV-2 foi estudado e sabe-se que ele compartilha cerca de 80% da identidade com o do SARS-CoV e apresenta 96% de similaridade ao coronavírus de morcego BatCoV RaTG13. Além disso, ambos compartilham ~ 76% de identidade de aminoácidos codificados.
Estudos anteriores demonstram que o SARS-CoV apresenta a glicoproteína spike (proteína S) em sua superfície e esta é responsável pelo reconhecimento do receptor celular humano e a sua fusão na membrana citoplasmática.
Pesquisas recentes relatam que durante a infecção viral tanto pelo SARS-CoV já conhecido, quanto pelo SARS-CoV-2 (novo coronavírus), apresentam o mesmo mecanismo de entrada na célula hospedeira. Eles utilizam como receptor a enzima conversora de angiotensina 2 (ECA2) para entrada e a serina protease TMPRSS2 para a iniciação da proteína S (figura 1).
A proteína S trimérica é clivada nas subunidades S1 e S2. As subunidades S1 são liberadas na transição para a conformação pós-fusão apresentando o domínio de ligação ao receptor (RBD), que se liga diretamente ao domínio peptidase (PD) da ECA2 (figura 2).
Após a fusão do vírus e entrada do material genético, o ciclo de vida viral continua dentro da célula para posterior replicação.
Siga nossas redes | Instagram | Facebok |
Referência/fonte
Yan, R et al. Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2. Science: 1444-1448. 2020. doi.org/10.1126/science.abb2762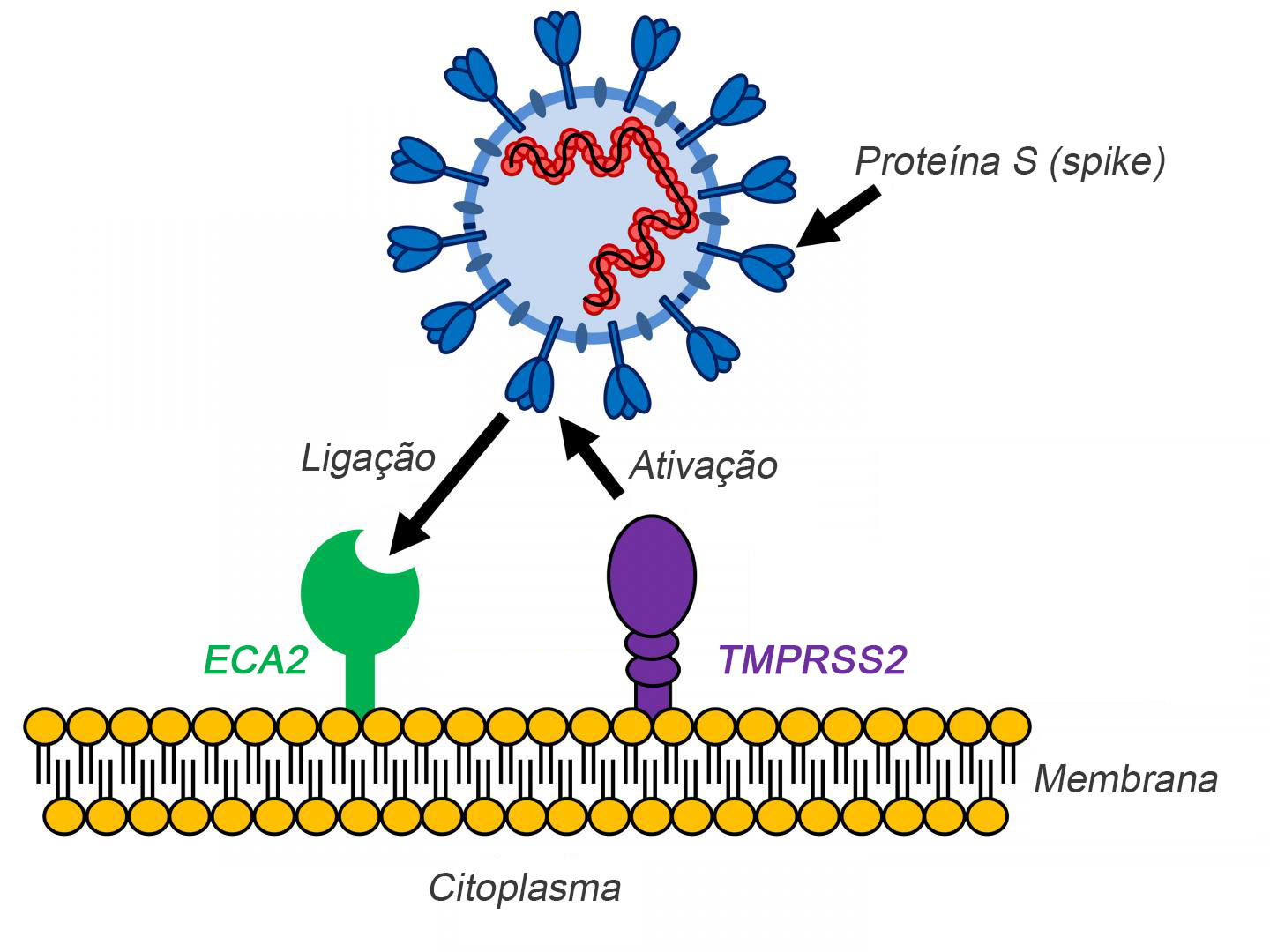 |
| Figura 1: representação do SARS-COV-2 ligando-se à célula. |
 |
| Figura 2: representação da ligação das subunidades à ECA2 |
Siga nossas redes | Instagram | Facebok |
Referência/fonte
M. Hoffmann et al. The novel coronavirus 2019 (2019-nCoV) uses the SARS-coronavirus receptor ACE2 and the cellular protease TMPRSS2 for entry into target cells. bioRxiv 2020.01.31.929042. https://doi.org/10.1101/2020.01.31.929042









0 comments